新闻中心 
单细胞转录组 数据分析教程 (Scanpy 篇)(单细胞转录组图谱怎么看)
最近一直在研究单细胞转录组数据分析,尤其是无监督条件下的scRNA-seq 数据分析方法,因此特地分享这个教程。目前scRNA-seq数据有开发比较成熟的工具,其中以Seurat 为代表。Scanpy 也是Seurat的python版本,一经发布就收到了广泛的关注,目前scanpy 和 Seurat 已经成为了scRNA-seq数据分析的几乎必备的工具。
我们以Scanpy 发布的 处理3k PBMC的数据为例子,来展开说一说,scanpy 在处理pbmc数据的时候每一步到底是在干什么,以及有什么用处。
scRNA-seq 数据到底长什么样子?
在详细的介绍scanpy之前,我们先来了解单细胞转录组也就是scRNA-seq 数据到底长什么样子。
Anndata 数据格式
如果想用好scanpy,一定首要清楚scanpy的数据存储结构Anndata。所有scanpy的function以及结果都会存在这个Anndata中。
Anndata的Tutorial,放在这里,计算机背景比较强的同学可以直接看这个doc就可以,但是还有很多生物方向的同学,对计算和数据结构并不是那么擅长,为此这里也简单解释一下。
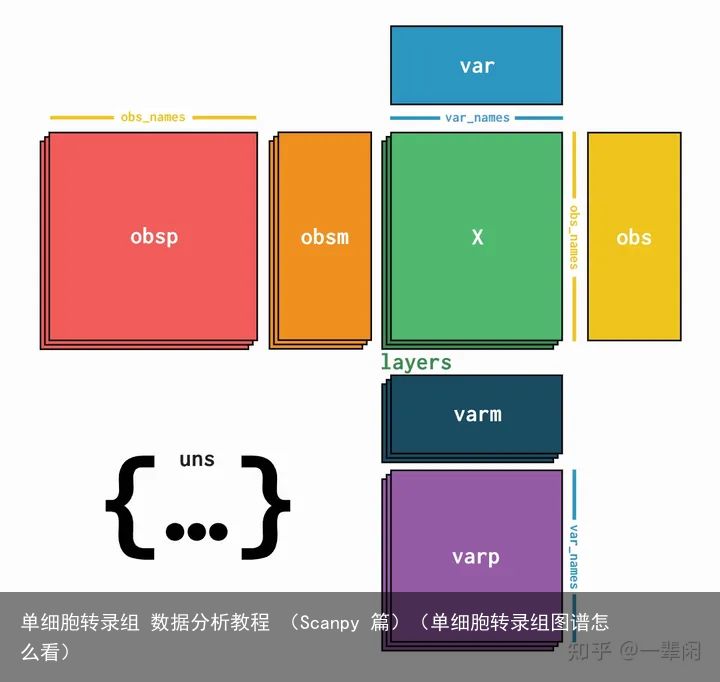 Anndata 数据结构
Anndata 数据结构AnnData诞生的原因
目前python 存储数据格式的形式已经有很多了,普遍来说numpy和dataframe已经是python中特别常用的数据矩阵格式,这时候为什么还需要有Anndata呢?最主要的原因就是单细胞转录组数据过于庞大与稀疏,采用Dataframe这种结构化储存的格式势必会对机器内存带来挑战。而AnnData采取的是scipy中稀疏矩阵存储的方式,大大节约了内存。
同时单细胞数据因为下游任务的要求,会对细胞或者基因进行注释和计算,而这些注释和计算的结果则对下游分析也有着一定的影响,为此除了单纯的稀疏矩阵存储单细胞转录组数据,AnnData也在数据中加入对细胞和基因以及其他辅助数学分析的量(var,obs,uns)来很好的解释单细胞数据。
obs
对细胞的注释,单细胞数据集一般是在无监督(没有标签的背景下分析的),为此我们需要对细胞进行注释,比如通过聚类,比如通过其他的方式(多模态表面蛋白标注)。因此Anndata.obs则是对每个细胞的标注,是一个Dataframe数据结构。
var
对基因的注释,我们同时某些基因也非常关注,比如某些基因是否是marker基因,某些基因是否是线粒体基因等等,这些对基因的注释都存储在Anndata.var中,也是个Dataframe的数据格式。
obsm
对细胞可视化的注释。我们知道单细胞数据集是多基因的高纬度数据集,因此如果想要进行可视化,就必须进行降纬操作,常用的降低维度的手段有PCA,TSNE,UMAP,通过这些降维手段后每个细胞都在embedding(降低后的维度)有个坐标信息,这个Anndata.obsm则是储存这个obs的坐标信息的。这是一种类字典的AxisArrays数据结构形式。可以采用字典的形式进行访问和查询。
varm
对基因的降维度操作,变量/特征的多维注释(可变结构化 ndarray)。为每个密钥存储长度n_vars的二维或更高维 nd数组。使用数据和变量进行切片,但行为类似于映射。一般来讲存储的是高维度基因降维后的PC值。同样也是可以采用字典的形式进行访问和查询。
obsp
对细胞间距离的注释。对于无监督背景下的数据分析,我们关心的是细胞之间的距离相似性,通过相似性可以对细胞进行聚类,而obsp存储的就是细胞之间的distance和通过采用流型学习,细胞之间建图的信息。(具体原理在稍后的文章进行阐述)
uns
非结构的注释。有时候我们不仅仅需要对细胞和基因进行注释,同时也需要存储一些其他的分析量。比如寻找差异基因,比如看差异基因的差异程度pvalue值等,这些非结构的注释一般都是存储在Anndata.uns中。这是一个Dict数据结构。
持续更新..








![[众诚云网科技]](/uploads/allimg/20190305/c4b08346cbe8b0efae6b132139c2d72a.png)
 2023-11-04
2023-11-04 浏览次数:次
浏览次数:次 返回列表
返回列表